Resumen
Para la vialidad celular, la rápida adaptación a la tonicidad extracelular es esencial y, junto con ello, la activación de una gran variedad de mecanismos celulares protectores del estrés osmótico. Recientemente se ha caracterizado TRPM3 (Transient Receptor Potencial Melastatin 3) (1). Este canal media la entrada de calcio en células epiteliales, luego de la aplicación extracelular de soluciones hipotónicas (2). Gracias a la modulación de este canal, las células epiteliales regulan su concentración iónica intracelular para adaptarse a las condiciones de su entorno y facilitar sus funciones celulares. La diversidad funcional de los canales iónicos se ha incrementado gracias a las variantes de splicing y a la heteromerización de subunidades regulatorias y formadoras del poro. En la familia de los TRPs, se han identificado muchas variantes de splicing, algunas de ellas, resultan en la carencia de respuestas a estímulos clásicos y otras modifican las propiedades de conducción. Una tercera posibilidad, consiste en ejercer un efecto dominante negativo, debido a la generación de un incremento exagerado en las concentraciones de calcio intracelular. TRPM3, presenta un gran número de diferentes variantes. Las primeras seis, fueron llamadas hTRPM3a-f (2), sin embargo, sólo se han reportado datos funcionales para hTRPM3a, el cual, excepcionalmente permite la entrada preferencial de cationes monovalentes, mientras que por el contrario, hTRPM3b presenta una selectividad por cationes divalentes, similar a la forma canónica. Además, se ha reportado una variante que presenta un total de 1325 residuos ya que su carboxilo terminal es considerablemente más corto (3). El amino terminal de esta variante, sin embargo, presenta 153 aminoácidos adicionales. En este último tiempo, se ha descrito cinco nuevas formas de TRPM3, TRPM3 1-5 (4). Estas, presentan un largo que va entre los 1699 – 1721 residuos aminoacídicos y además son caracterizadas por un amino terminal previamente desconocido de 61 residuos.
Considerando la relevancia del canal TRPM3 en la fisiología epitelial y el gran número de formas que éstos presentan, se piensa que un cambio en la tasa de variantes de splicing generada por diversos factores (5,6) como diferencias en pH, cambios osmóticos o choques térmicos, podría modificar el funcionamiento de las células epiteliales. Así, gracias a los antecedentes recogidos y fundados en la activación dependiente de la osmolaridad de estos canales, es probable que tanto TRPM3 como sus respectivas variantes funcionales, actúen como putativos osmosensores celulares.
Introducción
La secuencia codificante de los genes eucarioticos (exones), son típicamente interrumpidos por muchas secuencias largas no codificantes (intrones) que son removidas del RNA durante el proceso de splicing para dar lugar al mRNA. Muy a menudo, estos eventos ocurren cuando el RNA aún se está transcribiendo, momento en el cual, se remueve un intrón y se unen los exones adyacentes, para dar un producto de RNA más corto (7).
La existencia de los intrones facilita la recombinación de exones para producir diferentes proteínas con nuevas funciones, más aún, existen células que utilizan splicing para controlar su expresión génica (8). Durante algún tiempo, se pensó que la complejidad de un organismo estaba dada por el tamaño del transcriptoma, sin embargo, hoy se sabe, que a pesar del bajo número de genes predichos en nuestro genoma (30.000) los procesos postranscripcionales y postraducionales, tienen una enorme importancia al momento de explicar la variabilidad funcional.
El splicing alternativo es un proceso multifactorial complejo, regulado tanto por la interacción de factores tales como proteínas ricas en serina-arginina (proteínas SR) y ribonucleoproteínas heterogéneas (proteínas hnRNP) con sus secuencias blanco en el pre-mRNA (enhancer y represores del splincing) así como también, por la velocidad de transcripción de la RNA polimerasa II (9). El splicing alternativo también actúa en respuesta a estímulos externos como pH, temperatura, metales iónicos y condiciones osmóticas, entre otros (10).
La maquinaria de Splicing
El splicing es un proceso que químicamente consiste de dos reacciones sucesivas de transesterificación. El corte y el empalme son dependientes de la hidrólisis de ATP y son catalizados por un gran complejo de ribonucleoproteínas llamado “spliceosoma”. Este proceso dará como resultado la formación del mRNA, que será transportado al citoplasma y en donde será traducido a proteína en el caso de que ocurra splicing constitutivo o bien, a diversas proteínas en el caso de que se lleve a cabo splicing alternativo. Este evento, se lleva a cabo en el núcleo, aunque hay reportes de que también puede ser citoplásmico (11).
El spliceosoma, es un largo complejo proteína - RNA, el cual consiste en 5 tipos de RNAs nucleares pequeños (snRNA) y más de 50 especies de proteínas, las que se combinan para formar las ribonucleoproteínas pequeñas (snRNP)(12).
Durante el splicing, el spliceosoma reconoce secuencialmente tres posiciones en el RNA a través de un apareamiento de bases con el snRNA, que corresponde a los dinucleótidos GU y AG en los lugares de unión exón-intrón (sitios de splicing 5’) e intrón y exón (sitios de splicing 3’), respectivamente. El punto de empalme (branchpoint) es en general una adenina y mantiene una secuencia consenso. Entre el sitio de empalme y el sitio del dinucleótido AG, se encuentra una región rica en pirimidinas (uracilos, usualmente) llamada la región Py. En el primer paso de transesterificación, el grupo OH de la ribosa del residuo de adenina en el sitio de empalme, realiza un ataque nucleofílico hacia el sitio de splicing 5’, generando una ruptura del enlace fosfodiéster. Al mismo tiempo, la guanina del dinucléotido realiza un enlace a través de su extremo 5’ con el lado 2’ del residuo de adenina en el sitio de empalme. Después ocurre la segunda reacción de transesterificación, en la cual el grupo OH del extremo del exón realiza un ataque nucleofílico hacia el sitio de splicing 3’. Como resultado, se libera el intrón como una estructura en forma de lazo (lariat), al mismo tiempo que se unen los exones para generar el mRNA maduro. El intrón en forma de lariat se rompe nuevamente en su enlace 5’-2’ y se lineariza, posibilitando que sea degradado o bien procesado nuevamente para dar lugar a un RNA pequeño nucleolar rico en uracilo (UsnoRNA) (13) (Figura 1).
Figura 1. A) Flujo de la información genética. B) La reacción del splicing incluye dos pasos de transesterificación, en donde se requiere de ATP. En la reacción se libera el intrón y se ligan los exones.
- Los RNA pequeños nucleares cuyas longitudes oscilan entre 60 y 300 nucleótidos y son ricos en residuos de uracilo (UsnRNAs). Los cinco tipos son: el U1snRNA, el U2snRNA, el U4snRNA, el U5snRNA y el U6snRNA. El U3snRNA participa en eventos de maduración del pre-rRNA (RNA ribosomal inmaduro o precursor). Estos RNAs son transcritos por la RNA polimerasa II, salvo el U6snRNA, que lo transcribe la RNA polimerasa III. En el núcleo, los UsnRNAs se asocian con proteínas para formar ribonucleoproteínas pequeñas nucleares (UsnRNPs).
- Las partículas ribonucleoproteícas heterogéneas (hnRNPs) que contienen RNA heterogéneo nuclear (hnRNA), son estabilizadoras, es decir, su función radica en asociarse al pre-mRNA para impedir la formación de estructuras secundarias dadas por el apareamiento de bases. De ésta manera el pre-mRNA permanece accesible para interactuar con otras moléculas.
- Las proteínas SR (proteínas con dominios ricos en Serina y Arginina) representan una familia de factores de splicing que se reporta en vertebrados, invertebrados y plantas.
La secuencia de aminoácidos en estas proteínas es rica en residuos de serina (S) y arginina (R) en el extremo carboxilo, formando el dominio SR. Estas proteínas tienen varios papeles reguladores en el splicing como son la definición de los sitios de splicing y el acercamiento de las regiones de los dinucléotidos. Estas proteínas nucleares de 20 a 75 kDa, tienen capacidad de fosforilarse y se subdividen en dos tipos, las de Tipo I, presentan un único Motivo de Reconocimiento a RNA (RRM) en el extremo amino-terminal y su dominio SR carboxilo Terminal, y las de Tipo II, las cuales presentan dos Motivos de Reconocimiento a RNA y un dominio carboxilo terminal SR. Además, existen otras proteínas que pueden o no tener una estructura distinta del Motivo de Reconocimiento a RNA.
En las células de mamíferos se han identificado y aislado componentes esenciales del splicing, tales como el factor SF2/ASF, el factor SC35 y el 9G8, los cuales han sido identificados por medio de anticuerpos monoclonales (14).
La figura 2 ilustra cómo los factores involucrados en este proceso, interaccionan con el pre-mRNA de manera secuencial, formando complejos ribonucleoproteicos transitorios que permitirán que ocurran los dos pasos de transesterificación. En el primer paso se forma el complejo E, que se caracteriza por: la unión del U1snRNA de la U1snRNP al sitio de splicing 5’; este sitio de reconocimiento, está dado por:
- Una secuencia complementaria de U1snRNA con el sitio de unión exón-intrón, provocando un apareamiento con los primeros seis nucleótidos del intrón, delimitando así el exón 1;
- La unión del factor U2AF (que tiene el dominio SR) con el sitio Py;
- La participación en el complejo de los factores SC35, SF2/ASF, SF1/BBP y el U2snRNP.
Figura 2. Biología Molecular del splicing. El splicing ocurre de manera secuencial en varios pasos en el spliceosoma (complejos E-->A-->B1-->B2-->C1-->C2). Los factores no snRNPs (U2AF65/35, verde), proteínas SR, (amarillo) - ambas con el dominio RS- y los factores snRNP (naranja), participan en el reconocimiento y acercamiento de los sitios de unión intrón-exón, para que ocurran los dos pasos de transesterificación.
Posteriormente, el complejo E se transforma en el complejo A, cuando el U2snRNP se une al sitio de empalme. El complejo B1 se forma luego, cuando se asocia el trímero U4/U5/U6snRNP pre-ensamblado. Así, el U5snRNA se une al sitio de splicing 5’ y el U6snRNA se une al U2snRNA. Este complejo forma un spliceosoma maduro. Más tarde, se forma el complejo B2, en donde se libera el U1snRNA, el U5snRNA se mueve del exón hacia el intrón y el U6snRNA se une al sitio de splicing 5`. Este paso se lleva a cabo mediante un apareamiento de aproximadamente 20 bases entre el U1snRNA y el U4snRNA. Como consecuencia, se activa el spliceosoma para llevar a cabo su catálisis. La función de U5snRNA es interactuar con secuencias del exón, en el sitio del procesamiento 5´y 3´. El complejo C1 se forma cuando se libera el U4snRNA y el U5snRNA se une al exón en el sitio de splicing 3’. En este punto se cataliza la transesterificación, es decir, se rompe el sitio de splicing 5’ y se forma el lariat. Finalmente se forma el complejo C2, que contiene el trímero U2/U5/U6snRNAs, el cual permite el rompimiento del sitio de splicing 3’ y se ligan los exones (15). A pesar de los mecanismos descritos, la selección de los sitios de splicing, no están libres de error. Estas secuencias reguladoras del sitio de splicing y el spliceosoma, son incapaces de distinguir entre dos o más apareamientos alternativos de los sitios de splicing 5’ y 3’. Sin embargo, asisten el proceso de reconocimiento por unión a las proteínas regulatorias (16).
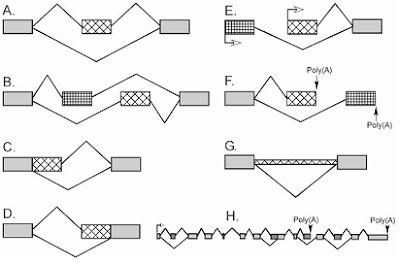
En un típico mRNA multiexón, el modelo de splicing alternativo puede ser alterado por muchas rutas, como lo muestra la figura 3. La mayoría de los exones son constitutivos, lo que quiere decir que ellos son siempre incluidos en el mRNA final. Un exón regulado, el cual algunas veces es incluido y otras excluido del mRNA, es llamado un exón casete. De esta forma, un exón mutuamente exclusivo ocurre cuando dos o más exones casete adyacentes son cortados y empalmados, incluyéndose sólo uno por vez. Los exones también pueden ser alargados o acortados por alteración en la posición de uno de sus sitios de splicing.
Alteraciones en promotores y sitios poli(A) alternativos cambian el sitio 3’ o 5’ de muchos exones.
Finalmente, algunos eventos son controlados por la falla en la remoción de un intrón, un modelos de splicing llamado “retención de intrón”. Un pre-mRNA puede exhibir múltiples sitios de splicing alternativo, usando diferentes modelos de inclusión, generando de esta forma, diferentes mRNAs. Cambios en la elección del sitio de empalme puede generar efectos en la proteína que será sintetizada. Pequeños cambios en la secuencia polipeptídica pueden alterar la unión de ligandos, actividad enzimática, regulación alostérica o localización de la proteína.
El hallazgo de TRPM3 en humanos y sus respectivas variantes
Durante el año 2003, Lee y colaboradores, encontraron un nuevo miembro de la familia TRP en humanos, el que fue llamado hTRPM3. Esta proteína está formada por un total de 1555 aminoácidos y posee las siguiente formas típicas de un canal TRP: seis dominios transmembrana, un dominio formador del poro, ubicado entre los aminoácidos 748-959, un motivo, conocido como la dominio TRP compuesto por la secuencia XWKFXR localizada río abajo del sexto segmento transmembrana y un domino coiled coil ubicado en el carboxilo terminal, río abajo de la dominio TRP. Un análisis de la secuencia genómica de hTRPM3, muestra que la región codificante alcanza 311 kb y comprende 24 exones separados por una de las secuencias intrónicas más largas del cromosoma 9. El gen de esta proteína se encuentra ubicado en el cromosoma 9q21.12 mientras que en ratas se localiza en el cromosoma 1q15 y en ratón en el 19. hTRPM3 es muy similar a hTRPM1 con un 57% de identidad y un 67% de similitud, además, se observó una fuerte homología (sobre el 80%) en el N-terminal, entre los aminoácidos 1 y 1219.
hTRPM3 fue originalmente detectado por medio de análisis de bioinformática, utilizando programas como, CLUSTALW, con el cual se realizaron alineamientos múltiples de secuencias correspondientes a los miembros de las familias de TRP conocidos, analizando la homología entre ellas y detectando los sitios más conservados, los cuales fueron utilizados para diseñar los partidores y así poder, más tarde, amplificar fragmentos a partir de una librería de cDNA de riñón humano. Los fragmentos resultantes, fueron ensamblados para generar el cDNA de largo total. Posteriores análisis de RT-PCR y Northern blot, determinaron la presencia de hTRPM3 en numerosos tejidos como cerebro, testículos, y de manera muy abundante, en riñón, sin embargo, parece estar ausente en una gran variedad de otros tejidos humanos. Esta última estrategia, se realizó, por medio de sondas específicas contra hTRPM3 de 645 pb.
Además, en el mismo estudio, se identificaron seis variantes desde una librería de cDNAs de riñón humano, usando partidores diseñados desde la secuencia codificante del gen hTRPM3.
Estas fueron designadas como hTRPM3 a-f. Sus largos van desde 1544 a 1579 aminoácidos (figura 4) (1,17), motivo por el cual no fue viable detectar estas variantes por el método de northern blot. Por otra parte, sólo se pudieron obtener datos funcionales de hTRPM3a, la cual, excepcionalmente permite la entrada preferencial de cationes monovalentes, y de hTRPM3b, cuya expresión heteróloga, exhibe la entrada de calcio y magnesio dependiente de la concentración, siendo por lo tanto, un canal selectivo a cationes divalentes, el cual además es bloqueado por cationes monovalentes, de manera similar a la forma canónica.
La variante de splicing hTRPM3a presenta la exclusión del exón 7 y 11, lo que genera una deleción del N terminal citoplasmático. Luego, la variante hTRPM3b además de presentar una exclusión del exón 7 y una deleción entre el exón 11 y 12, muestra una inserción de 12 aminoácidos entre el exón 13 y el 15, lo que genera una deleción y una posterior inserción en el N citoplasmático. hTRPM3d, presenta la exclusión del exón 7, 11y 13 y presenta una deleción entre el exón 14 y15, esto provoca deleciones en el N-terminal citoplasmático, tampoco presenta datos funcionales. Por su parte, la variante hTRPM3e, presenta la exclusión del exón 7 y 13, además de una inserción de 12
aminoácidos entre el exón 14 y 15 y una deleción entre los exones 11 y 12, esto también genera deleciones e inserciones en el N-terminal citoplasmático (sin datos funcionales). La variante hTRPM3c presenta una exclusión del exón 7 y del exón 11, además de una inserción de 12 aminoácidos en el exón 20 lo que genera una deleción del amino terminal citoplasmático y por otro lado, variaciones en la región del poro del canal. El gen hTRPM3f incluye el exón 4, ausente en todas las demás variantes y además presenta 18 aminoácidos codificados por el exón 8. Por último, se encuentra la isoforma 10, la cual sólo es encontrada en cerebro y riñón de humanos fetales (2). Esta variante presenta la exclusión del exón 7 y además la incorporación de 6 aminoácidos en el último exón, lo que genera un canal de Ca2+ que modula su entrada por una alteración en la osmolaridad extracelular.
Figura 4: Variantes de splicing de hTRPM3. Se detectaron cinco variantes adicionales: b-f y KIAA1616. Algunas de las diferencias en las variantes b-f son mostradas en un cuadro. Los exones codificantes son mostradas por números que van desde el 1 al 24. Como se muestra en la figura, el gen hTRPM3b contiene un exón extra, el exón 11. el gen hTRPM3c contiene un exón, que corresponde al 20, el cual produce la adición de 12 aminoácidos a la proteína . El gen hTRPM3d el exón 13 es excluido, mientras que en el gen hTRPM3e incluye el exón 11. Finalmente, el gen hTRPM3f incluye el exón 4 y 18 aminoacídos codificados por el exón 8.
Por su parte Grimm y colaboradores (2), usando la estrategia de PCR 5’ y 3’ RACE, identificaron 3 fragmentos de cDNA desde cerebro fetal y riñón, a partir de lo cual predijeron una proteína de 1325 aminoácidos, que presenta un carboxilo terminal considerablemente más corto que el presente en la forma canónica, sin embargo, tiene un amino terminal de 153 aminoácidos adicionales, el cual no forma parte de ninguna otra variante de TRPM3. Posteriores análisis de electrofisiología revelaron que esta proteína correspondía a un canal catiónico no selectivo, permeable a Ca2+ y sensible a la osmolaridad del medio extracelular y la llamaron TRPM31325. La actividad de los canales TRPM31325 es reportada por aumentar el volumen celular en soluciones hiposmóticas.
La información disponible de los transcritos revela que muchas diferentes especies de mRNA codifican para una gran variedad de proteínas TRPM3. La variabilidad de estos transcritos depende de dos o probablemente de tres posiciones de inicio y de dos diferentes sitios de término. La diferencia en el término del C-terminal depende de un sitio de splicing altamente conservado que se encuentra en las variantes largas en el aminoácido 1318. En mRNAs que codifican para variantes de alto peso molecular, la maquinaria de splicing ignora este sitio, mientras que en especies de mRNA que codifican para variantes de menor peso molecular, el resultado del reconocimiento de este sitio es la adición de un exón accesorio que codifica sólo para 8 aminoácidos.
La actividad constitutiva de TRPM3 de ratón, también fue caracterizada por Oberwinkler (4). Ellos describen TRPM3 como un canal proteico de 1725 aminoácidos, formado por 28 exones y que alcanza 850 kb. Este canal es permeable a calcio y magnesio. Adicionalmente, ellos analizan una gran variedad de variantes de splicing generadas en cerebro de ratón, las que además, confirman las variantes encontradas por Lee (18). Este grupo, es responsable de caracterizar una variante con 10 aminoácidos adicionales en el asa extracelular siguiente al putativo segmento transmembrana número 6. La presencia de los aminoácidos adicionales resulta en una pérdida de permeabilidad iónica. De esta manera, cuando la variante y la silvestre se expresan juntas, la permeabilidad es disminuida, lo que llevaría a pensar que esta variante de poro de TRPM3 presenta un carácter dominante negativo. Análisis detallados en un futuro, podrían mostrar si TRPM3 poro-variante es traducida sola o junto con otras variantes de TRPM3 en célula única. La traducción de TRPM3 poro-variante junto a otras variantes de TRPM3 pueden ayudar a la célula a modular su permeabilidad iónica celular a nivel traduccional.
Las variantes identificadas por Oberwinkler, fueron designadas como TRPM3a1, TRPM3a2, TRPM3a3, TRPM3a4 y TRPM3a5. Su secuencia aminoacídica comprende desde 1699 a 1721 aminoácidos, los cuales son codificados por los exones 1, 3-7 y 9-28. Los exones 2 y 8 no están presentes en el cDNA de variantes de TRPM3 de ratón, pero 18 aminoácidos codificados por el exón 8 han sido detectados recientemente en variantes como el hTRPM3f (18). El exón 2 codifica una secuencia de 59 aminoácidos que corresponde a la parte amino terminal de la variante TRPM31325 en humanos (2) pero está ausente en las variantes a - f de hTRPM3. El exón 1 codifica para una secuencia amino terminal completamente nueva que no está presente en alguna de las variantes de TRPM3 humanos descritos.
Se ha visto que las proteínas TRPM3 de ratón muestran aproximadamente un 97% de identidad en su secuencia aminoácidica con las variantes humanas. Así el TRPM3a1 de ratón corresponde a la humana TRPM3c, mTRPM3a2 a hTRPM3a, mTRPM3a3 a hTRPM3b, mTRPM3a4 a hTRPM3e y mTRPMa5 a hTRPM3d. TRPM3a1 y TRPM3a2, difieren solo en una región entre el quinto y el sexto segmento transmembrana, donde se reporta la región formadora del poro del canal. Esta región es 12 aminoácidos más largo en TRPM3a1 comparado con TRPM3a2, adicionalmente, una prolina es sustituida por una alanina, por lo tanto, presentaría una mutación puntual en el exón 20. Estos datos, muestra que TRPM3 está sujeto a frecuente splicing alternativo, implicando que diferencias funcionales descritas pueden ser causadas por las diferentes estructuras primarias de las variantes. Además, la fuerte correspondencia entre los respectivos transcritos indica que no sólo las secuencia aminoacídicas si no también los eventos de splicing, son altamente conservados entre ratón y humano. Así, la fuerte conservación generalmente sugiere la importancia de las proteínas, como
en este caso de TRPM3, pero también muestra la importancia de su regulación y modificación por splicing alternativo.
Variantes de TRPM3 como reguladores osmóticos
Aún no se tiene claro, cuales son los factores que favorecen la presencia de una u otra variante del canal, sin embargo, es probable que esto se genere a modo de una regulación celular frente a ciertos cambios, como es el caso de una variación en el gradiente osmótico (19).
Diferencias en el gradiente de concentración iónico son responsables de la hinchazón o del encogimiento de la célula por incremento o disminución del agua intracelular, respectivamente (20). La viabilidad de las células depende de mecanismos que aseguren una rápida adaptación a la tonicidad extracelular. Por modulación de canales iónicos y transportadores, las células modulan su concentración iónica intracelular para adaptarse a las condiciones ambientales y para facilitar su función celular (3,21).
TRPM3 y sus variantes de splicing son proteínas que se encuentran específicamente a nivel de células epiteliales del túbulo colector, en el riñón. Estas, han sido caracterizadas, como proteínas que median la entrada de calcio en las células en soluciones extracelulares hiposmóticas (2,21). Se ha visto, que al menos dos variantes de este canal pueden ser expresados desde este
gen presentando selectividades iónicas completamente diferentes. El canal hTRPM3a, preferencialmente conduce la entrada de cationes monovalentes, mientras que hTRPM3b fuertemente favorece la entrada de cationes divalentes. Previamente, la variante humana TRPM31325 ha sido reportada por mediar el incremento en la entrada de calcio cuando la osmolaridad de la solución extracelular cambia desde 300 a 200 mosmol/kg, un estímulo que induce un considerable aumento de volumen de las células. In vivo, cada uno de los cambios en la selectividad iónica puede tener considerables consecuencias para la función del canal y la fisiología de las células que lo expresan.
Minigenes, una ventana al conocimiento de la regulación de splicing para TRPM3
Considerando la escasa información existente sobre la regulación del splicing alternativo del canal TRPM3 y dada la importancia que se les ha otorgado en el proceso de osmoregulación, es necesario conocer a cabalidad la regulación de este mecanismo y para esto, se pueden abordar preguntas básicas tales como, ¿cuáles son los elementos exónicos e intrónicos que favorecen o reprimen el splicing?, ¿cuáles exones son constitutivos y cuáles son alternativos?, el tamaño de los exones ¿es importante para preferir su inclusión?, ¿existe algún tipo de splicing alternativo, más favorecido que otros en estas célula epiteliales? y si es así, ¿qué elementos regulatorios, cis y trans, son necesarios para que esto ocurra?, entre muchas otras.
Para resolver alguno de estos problemas, es posible aplicar el uso de minigenes. Como su nombre lo indica, un minigen, es una versión simplificada y artificial de un gen (22), cuyo uso, nos permitirá:
(1) determinar el rol de los sitios de splicing 3’ y 5’, en el reconocimiento de un exón;
(2) identificar elementos exónicos e intrónicos, capaces de aumentar o reprimir el splicing,
(3) determinar si una variante alélica tiene algún efecto en la eficiencia del splicing,
(4) identificar elementos requeridos para la regulación específica de una determinada célula.
(5) identificar elementos requeridos para la regulación por factores activadores trans específicos (23).
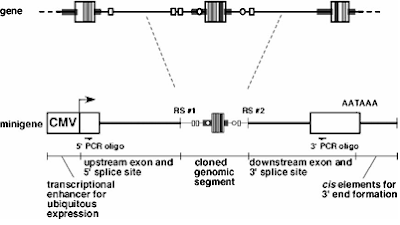
Como se observa en la figura 5, los minigenes, contienen un segmento genómico proveniente de un gen de interés que incluye una o varias regiones de splicing alternativo y regiones genómicas flanqueantes. El segmento genómico es generado por amplificación por PCR ya sea, usando como templado directamente el DNA genómico o un fragmento de DNA genómico clonado.
Además, es muy importante generar sitios para enzimas de restricción que permitan el posterior clonamiento del fragmento, en el plasmidio. El largo de las secuencias genómicas flanqueantes, dependerá de nuestro objetivo en el análisis y se puede encontrar en un rango desde decenas a cientos de nucleótidos. Para determinar si una variante alélica es responsable de alterar el splicing, o para probar si un motivo específico en la secuencia es el elemento responsable para un putativo factor regulatorio, el minigen podrá incluir los elementos cis de interés. Para identificar elementos cis activadores requeridos para la regulación celular-específica de un exón alternativo, se podrá cambiar aquel fragmento que probablemente contenga elementos regulatorios primarios. Basados en el conocimiento actual, los elementos regulatorios primarios se encuentran típicamente entre 200 - 300 nucleótidos río arriba y/ o río abajo de un exón regulado. Por otro lado, cuando un segmento genómico ha sido mostrado por contener elementos que son suficientes para regular el splicing, acortamientos de estas secuencias desde los sitios desde sus sitios 3’ o 5’, podrían ser usados para definir pequeños fragmentos genómicos necesarios y suficientes para su regulación. Esta estrategia, servirá para determinar el segmento genómico mínimo que facilita la identificación de un elemento cis importantes, gracias a la eliminación funcional de los mismos. Por otro lado, si esto fallara, sería recomendable utilizar segmentos genómicos largos, para demostrar regulación. Por ejemplo, un fragmento genómico que abarca desde un exón río arriba del exón alternativo a un exón río abajo del siguiente exón alternativo, es muy probable que incluya todos los elementos regulatorios relevantes y necesarios para su regulación.
De esta forma, el sistema de minigenes es una importante herramienta para la identificación y análisis in vivo de reguladores como elementos cis y factores trans, que establecen un splicing alternativo eficiente y regulado, además, la expresión de preRNAs mediante el sistema minigene, por transfección transciente, provee un rápido ensayo para conocer la pérdida de función y/o el análisis de la pérdida o ganancia de elementos cis y/o factores trans que afectan la regulación de la transcripción. Por lo tanto, el uso de minigenes, nos darán algunas luces sobre el mecanismo de regulación del splicing de estos canales y sobre el modo de regulación específicamente en células epiteliales.
Conclusiones
Recursos obtenidos desde base de datos genómicos ofrecen mayor facilidad a la identificación de muchos de los genes de TRP en los últimos 10 años por revisiones similares a las descritas. Sin embargo, ya que la regulación transcripcional y el procesamiento post transcripcional del mRNA son escasamente predecibles y muy complejos, se hace cada vez más necesario descifrar el transcriptoma del TRP. Los diferentes largos de los mRNAs de TRPM3 y las diferencias en la masa molecular de las proteínas, resultan de diferentes mecanismos de regulación de splicing.
Ciertamente, la información en el presente, acerca de cuáles son las posibles mecanismo de regulación del splicing para este canal, así como las implicancias funcionales que tengan cada variante a nivel celular y fisiológico, son muy escasas, sin embargo, estos antecedentes dan luces de la importancia del canal y sus variantes, siendo necesario estudiar en detalle los mecanismos que imperan en el procesamiento del RNA, estableciendo por ejemplo, en qué situaciones se activa una u otra variante, o determinar si se coexpresan in vivo con la proteína canónica, generando un efecto dominante negativo, como es el caso de la variante 1325.
Sin duda, aún queda mucho por investigar, sin embargo, es un camino obligado, para conocer en detalle el mecanismo de regulación del volumen de la célula a nivel molecular.
Referencias
1. Vázquez E, V. M. (2006) Semin Cell Dev Biol 17(6), 607-617
2. Grimm, C., Kraft, R., Sauerbruch, S., Schultz, G. and Harteneck, C. (2003) J. Biol. Chem. 278, 21493–21501
3. Grimm, C., Kraft, R., Schultz, G. and Harteneck, C. (2005) Mol. Pharmacol. 67, 798-805
4. Oberwinkler, J., Lis, A., Giehl, K.M., Flockerzi, V. and Philipp, S.E. (2005) J. Biol. Chem. 280, 22540-22548
5. Stamm, S. (2002) Hum Mol Genet 11, 2409–2416
6. Tarn, W. (2007) J Biomed Sci 14(4), 517-522
7. Black, D. L. (2003) Annu. Rev. Biochem 72, 291-336
8. Black, D. (2000) Cell 103, 367–370
9. Kornblihtt AR, d. l. M. M., Fededa JP, Munoz MJ, Nogues G. (2004) RNA 10(10), 1489-1498
10. S., S. (2002) Hum Mol Genet 11, 2409–2416
11. Eran, M., and T. Misteli. (2005) Splicing Misplaced. Cell 122, 317-318
12. Staley JP, G. C. (1998) Cell 92, 315–326
13. Brow, D. A. (2002) Annu Rev Genet 36, 333-360
14. Sun Q, M. A., Hampson RK, Krainer AR, Rottman FM. (1993) Genes Dev 7(12B), 2598-2608
15. Blaustein M, P. F., Srebrow A. (2007) Int J Biochem Cell Biol 39(11), 2031-2048
16. Smith CW, V. J. (2000) Trends Biochem Sci 25, 381–388
17. Annette Lis, U. W., Stephan E. Philipp. (2005) Naunyn-Schmiedeberg’s Arch Pharmacol 371, 315-324
18. Lee, N., Chen, J., Sun, L., Wu, S., Gray, K.R., Rich, A., Huang, M., Lin, J.H., Feder, J.N., Janovitz, E.B., Levesque, P.C. and Blanar, M.A. (2003) J. Biol. Chem 278, 20890-20897
19. Stamm, S. (2002) Human Molecular Genetics 11(20)
20. Reiter, C. H. a. B. (2007) Cell and Molecular Biology of TRP Channels 35(part 1), 91 - 95
21. Strotmann, R., Harteneck, C., Nunnenmacher, K., Schultz, G. and Plant, T.D. (2000) Nat. Cell Biol 2, 695-702
22. Baralle, D. B. a. M. (2005) J. Med. Genet 42, 737-748
23. Cooper, T. A. (2005) Methods 37 331–340
Para una forma más interactiva de entender el mecanismo de splicing, puedes observar el siguiente vídeo.




No hay comentarios:
Publicar un comentario